]服务简介
基因组测序是指通过使用高通量DNA测序和生物信息学来组装和分析整个基因组的功能和结构。利用基因组测序可以对个体变异类型进行分析或对群体进行差异性分析。de novo测序后利用信息学方法,还可获得物种的遗传图谱。
]服务优势
◆通量高、质量高、速度快:采用Illumina测序平台完成。在保证质量的同时,样本测序与分析速度比传统测序分析速度大幅提高;
◆定制化分析策略:根据不同测序物种和测序方案,定制化选择参考基因组版本、比对算法和注释用数据库区域信息等;
◆可测样本种类丰富:丰富的科研服务经验,目前可对gDNA、细胞、血液、新鲜组织、冷冻组织、石蜡切片(FFPE)、制备好的文库等多种类型的样品进行最佳方案设计及检测;
◆强大的组学联合分析能力:将基因组重测序与转录组测序和甲基化测序等技术进行结合,将单一的基因变异数据进一步拓展;
◆完善的售前、售后服务:经验丰富的技术支持和生物信息工程师提供研究策略指导及完善的售后服务。
]服务项目
基因组de novo测序
全基因组测序
外显子测序
目标区域测序
线粒体基因组测序
泛基因组测序
单细胞基因组测序
简化基因组测序(种质资源鉴定、群体进化、GWAS、图谱构建、BSA定位)
]服务流程
]样本量要求
样本类型 | 样本量及样本要求 |
动物和各类组织样本 | ≥0.5g |
植物组织样本 | ≥2g |
全血 | ≥1mL |
细胞 | 4×106个 |
DNA样本 | DNA总量≥2ug 样本浓度≥100ug/uL 样品纯度OD260/280为1.8~2.2 DNA主带清晰无明显降解 |
]技术参数
检测平台:全基因组测序采用先进的Illumina测序平台,快速、高效地读取高质量的测序数据;
合作伙伴的高性能计算平台(High Performance Computing,HPC)采用DELL计算节点和Isilon存储的高效组合,实现快速稳定的测序数据分析及交付;
测序深度:肿瘤:癌组织50X,癌旁组织/血液样品30X;
遗传病等其他样品:30X~50X;
服务周期:15~22个工作日。

]服务内容
全基因组重测序通过对个体进行全基因组测序,全面解读基因组上的变异信息,预测该变异信息与疾病的关联性。
]疾病基因组
标准信息分析:
1.数据质控:去除接头污染和低质量数据
2.与参考序列进行比对、统计测序深度及覆盖度
3.SNP/InDel/SV/CNV 检测、注释及统计
4.基因组变异 Circos 图展示
个性化分析
1. 药物效应多态性的遗传学机理研究使用 PharmGKB 和 Drugbank 数据库对药物基因组项目进行注释和分析,需客户提供所关注的药物名称。
2. 生存分析(基于临床随访数据)
3. 疾病显著性关联位点分析(建议基于 150 对以上 case/control)
4. 疾病显著性关联基因分析(建议基于 150 对以上 case/control)
备注:Control 的选择范围
5. HLA 分析(分析内容参照人 HLA 捕获测序分析内容)
6. 线粒体基因组分析(分析内容参照人线粒体基因组测序分析内容)
]高级信息分析(单基因病)
(一)基于变异有害性的筛选
1. 突变位点筛选
2.突变位点有害性分类(ACMG)
3.Non-coding 区突变位点筛选
4.结构变异 CNV/SV 有害性分析
(二)基于选样信息的筛选
1.显性/隐性遗传模式分析(需合作方提供家系图)
2.家系连锁分析(家系样本)
3.纯合子区域(ROH)分析(近亲结婚家系样本)
4.共有突变基因筛选(散发样本)
(三)基于基因功能和表型的筛选
1.候选基因功能富集分析
2.候选基因通路富集分析
3.候选基因与疾病相关性排序
]高级信息分析(复杂疾病)
(一)基于变异有害性的筛选
1. 突变位点筛选
2.突变位点有害性分类(ACMG)
3.Non-coding 区突变位点筛选
4.结构变异 CNV/SV 有害性分析
(二)基于选样信息的筛选
1.显性/隐性遗传模式分析(需合作方提供家系图)
2.新生突变筛选(核心家系)
3.共有突变基因筛选(散发样本)
(三)基于基因功能和表型的筛选
1.蛋白功能互作网络(PPI分析)
2.候选基因功能富集分析
3.候选基因通路富集分析
4.候选基因与疾病相关性排序
]癌症基因组学
基本信息分析
1.数据质控 : 去除接头污染和低质量数据
2.与参考序列进行比对、统计测序深度及覆盖
3.Somatic SNP / InDel / SV /CNV 检测、注释及统计 ( 成对样本 )
4.易感基因筛查
5. 高频突变基因统计及通路富集分析
6.NMF 突变特征及突变频谱分析
7.NovoDriver 已知驱动基因筛选
8. 基因组变异 Circos 图展示
高级信息分析
1. MRT 高频突变基因相关性分析
2. OncodriveCLUST 驱动基因预测
3. 突变位点分布情况分析(新)
4. 高频 CNV 分析
5. 融合基因检测及 Circos 图展示(新)
6. ABSOLUTE 肿瘤纯度及倍性分析
7. 杂合性缺失 (LOH) 分析(新)
8. 瘤内异质性及克隆结构分析
9. NovoDrug 高频突变基因靶向用药预测
10. NovoDR 耐药突变筛选
11. NovoNoncoding 非编码区高频突变分析
个性化分析
1.肿瘤进化树分析
2. NovoVirus 病毒整合分析
3. 克隆结构分析(新)
3.1 同一病人多区域取样克隆结构分析(EXPANDS)
3.2 多样本间克隆结构进化分析(Pyclone)
4. 临床数据整合
5. 肿瘤分型分析(新)
5.1 微卫星分析
5.2 重排特征分析
5.3 端粒长度分析
5.4 Kataegis 分析
6. 新抗原预测(新)
7. 驱动基因预测(新)
8. 突变频谱 3D 展示图(新)
9. Conpair 分析样本间一致性和污染程度(新)
测序数据的质量主要分布在Q30(≥80%)以上,这样才能保证后续分析的正常进行。测序深度指比对到参考基因组的碱基总数除以基因组大小。覆盖度指被测到的该物种基因组的碱基总数占该物种基因组长度的百分比。测序深度和覆盖度能够直接反应测序数据的均一性及与参考序列的同源性。
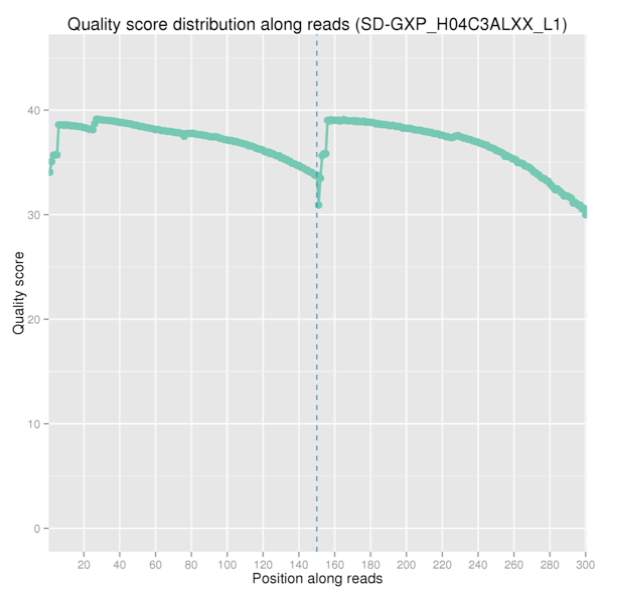

图1 数据质量分布 图2 测序深度
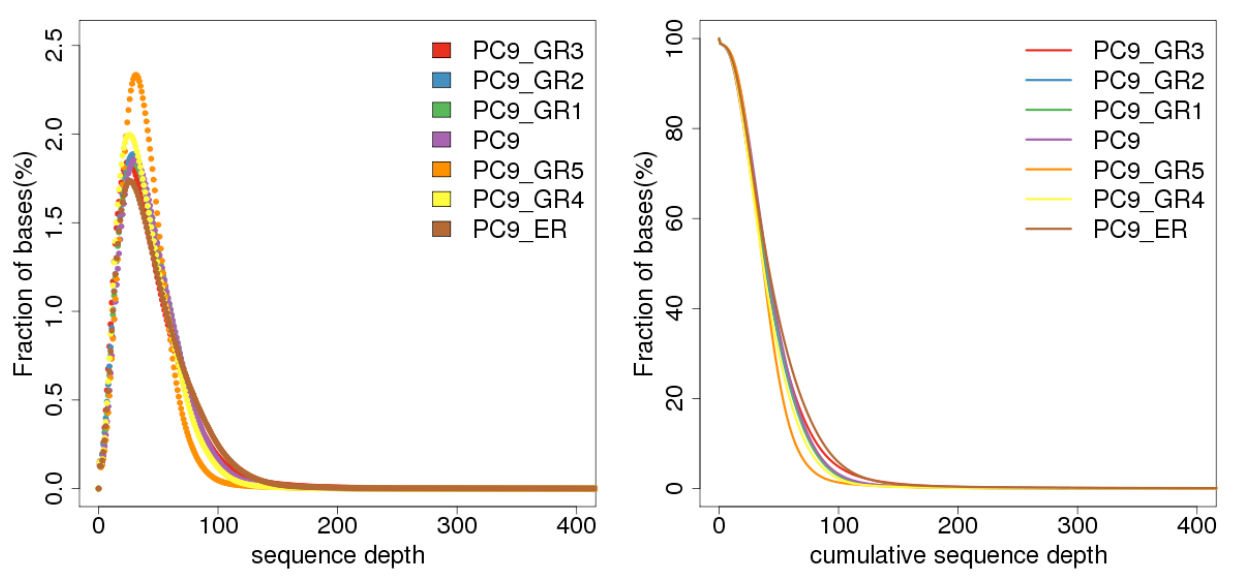
图3 样本每个染色体的平均覆盖深度柱形图和覆盖率折线图
CNV(拷贝数变异)是一类特殊的结构变异,指的是基因组上大片段序列拷贝数的增加或者减少,可分为缺失(Deletion)和重复(Duplication)两种类型,是一种重要的分子机制。CNV 能够导致包括癌症在内的复杂疾病,对于染色体水平缺失、扩增的研究已经成为肿瘤研究热点。
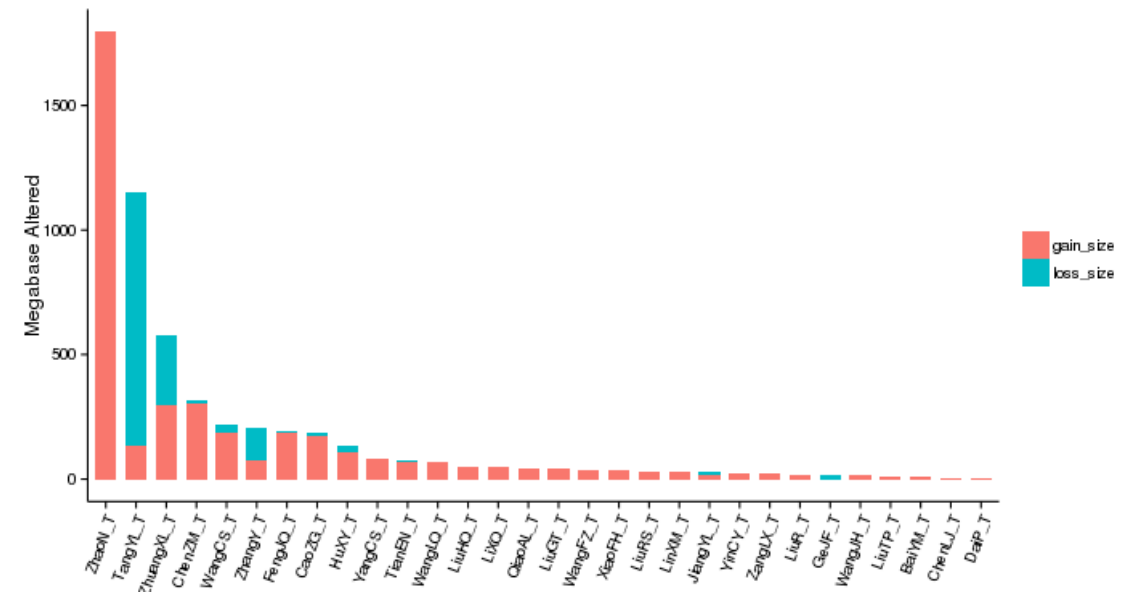
图4 Somatic CNV 的分布
通过突变频谱分析我们可以得知各个肿瘤样本各种类型突变的数量及样本是否有某种类型突变的偏好性。通过分析体细胞突变特征,可以研究不同癌种的体细胞点突变特点。
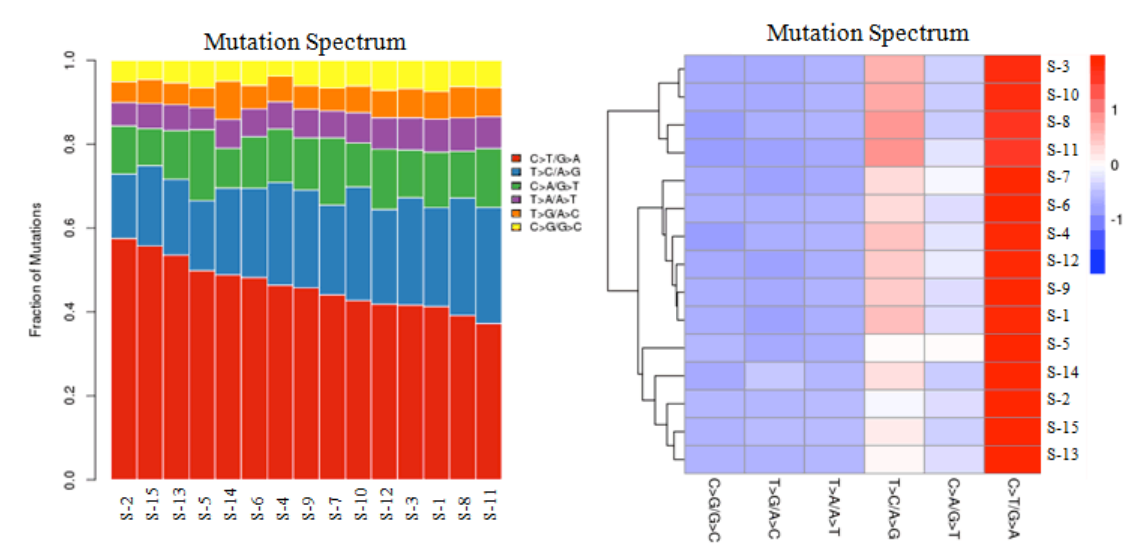
图5 体细胞 SNV 突变频谱柱状图和热图
在生物体内,不同基因相互协调行使其生物学功能,通过 Pathway 显著性富集能确定高频突变基因参与的最主要生化代谢途径和信号转导途径。
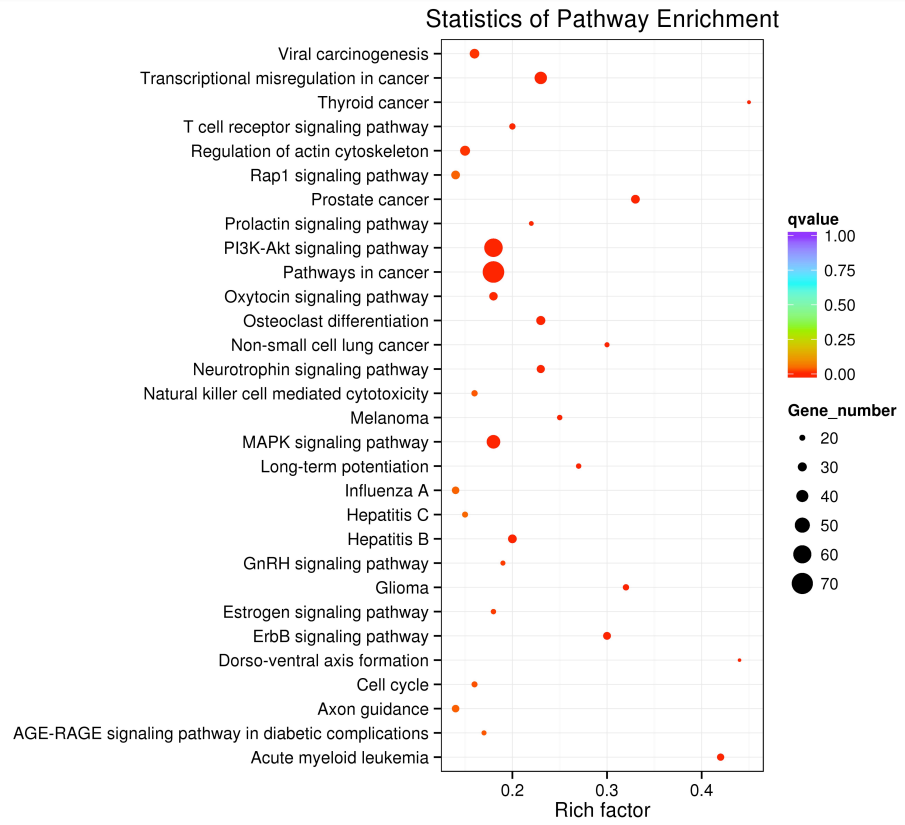
图6 显著富集通路展示图
根据检测得到的体细胞突变信息整合成 Circos 图,对基因组数据进行可视化展示。
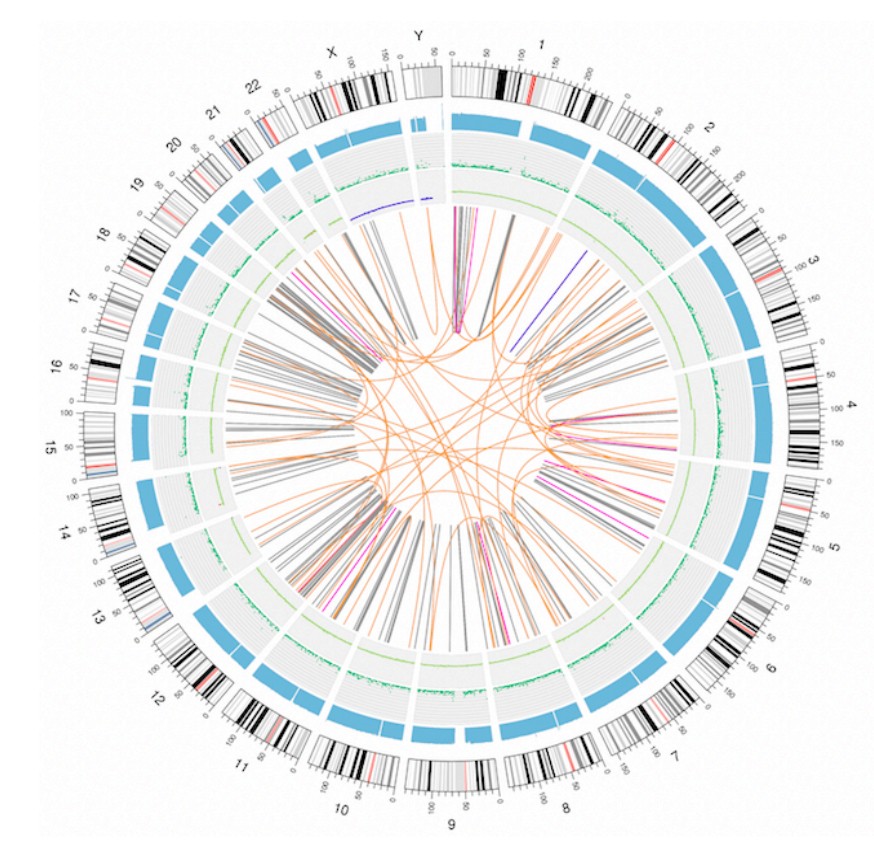
图7 体细胞变异 Circos 个性化展示图
高频突变基因(Significant mutation gene, SMG)在单个样本中可能存在协同(两 个基因倾向于同时发生突变)或者互斥(一个基因发生突变,另外一个基因突变概率大大降低)两种关系,协同作用实则说明两个基因可能必须同时突变才会产生致癌作用,互斥关系则认为单个基因发生突变足以诱发正常细胞向癌细胞的转化。
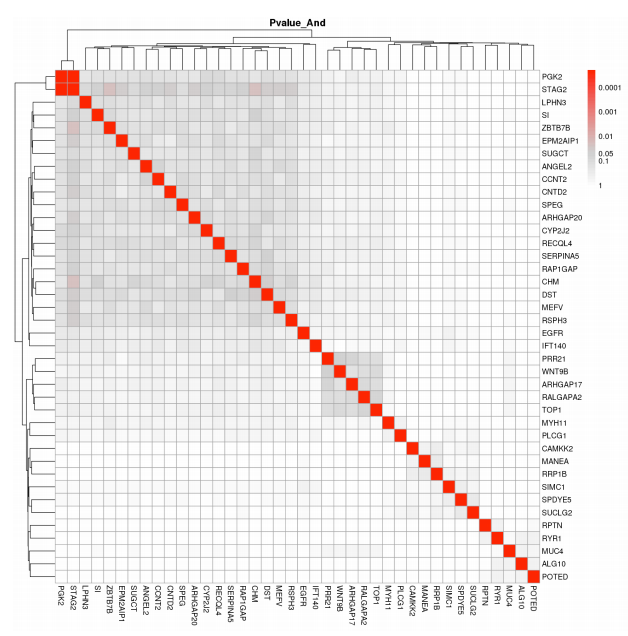

图8 高频突变基因协同作用与互斥作用热图
寻找肿瘤特异性的 Somatic CNV 不仅可以更好的理 解肿瘤发生的分子机制,也可以为开发有效的肿瘤治疗药物提供靶点。利用 GISTIC软件来评估样本集合中显著扩增或缺失的基因组区域,筛选出现频率较高的 CNV 区域。
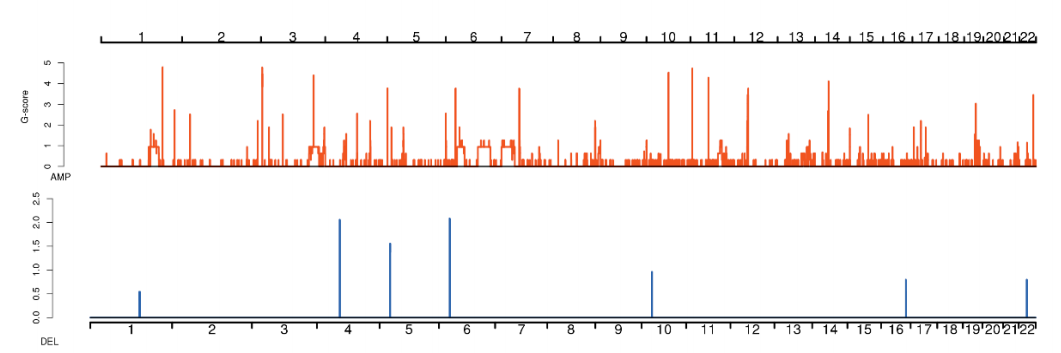
图9 高频 CNV 的分布图
相关文献
Choi SH, Weng LC, Roselli C, et al. Association Between Titin Loss-of-Function Variants and Early-Onset Atrial Fibrillation. JAMA. 2018;320(22):2354-2364.





