]服务简介
表观组学测序(Epigenomics Sequencing)是以高通量测序平台为基础,研究基因表达及调控的可遗传变化的技术。根据研究内容可将表观组学分为两类:DNA/RNA的化学修饰和DNA/RNA与蛋白质相互作用。常用的表观遗传学测序方法包括BS-seq和ChIP-seq。
全基因组DNA甲基化测序(Whole Genome Bisulfite Sequencing,WGBS)是将重亚硫酸盐处理方法和Illumina高通量测序平台相结合,对有参考基因组的物种在全基因组水平进行高精准甲基化研究。WGBS可以达到单碱基分辨率,精确分析每一个胞嘧啶的甲基化状态,从而构建精细的全基因组DNA甲基化图谱。
ChIP-Seq是将染色质免疫共沉淀(Chromatin Immunoprecipitation,ChIP)与二代测序相结合的表观遗传研究技术,能够高效地在全基因组范围内对DNA和蛋白的相互作用进行检测,通常用于转录因子结合位点或组蛋白特异性修饰位点的研究。
RIP-seq是研究细胞内RNA与蛋白结合情况,以RNA免疫共沉淀(RIP)为基础,采用特异抗体对RNA结合蛋白或者特殊修饰的RNA进行免疫共沉淀后,分离RNA,通过Illumina测序,在全转录组范围内研究被特定蛋白特异结合的RNA区域或种类,且可比较多个样品间差异。
]服务优势
◆定制化分析策略:根据不同测序方案和测序目标,定制化选择比对算法、Peak Calling算法以及注释用数据库区域信息。
◆ 高深度、高灵敏度:对低频甲基化位点的探测率高,对甲基化和非甲基化均等捕获;单碱基分辨率,可精准检测每个胞嘧啶的甲基化状态。
◆低成本、高效率:使用液体捕获技术,精准获得全基因组甲基化的C位点,大大提高捕获效率;同时靶标区域的选择策略可降低成本,得以实现多样本快速实验。
◆强大的组学联合分析能力:将ChIP-Seq与甲基化测序、转录组测序以及全基因组测序等技术进行结合,将单一的蛋白结合数据更进一步拓展。
◆定制化信息分析:针对不同平台的数据,结合客户的需求,灵活进行定制化信息分析。
◆完善的售前售后服务:经验丰富的技术支持和生物信息工程师提供研究策略指导及完善的售后服务。
]服务项目
甲基化测序
RRBS
Chip-Seq
RIP/DIP-Seq
]服务流程

]样本量要求
样本类型 | 样本量及样本要求 |
动物和各类组织样本 | ≥50mg |
植物组织样本 | ≥50mg |
DNA样本 | DNA总量≥50ng 样本浓度≥100ng/uL 样品纯度OD260/280为1.8~2.2 DNA主带清晰无明显降解 |
]甲基化测序技术参数
检测平台:基因组甲基化测序采用先进的Illumina测序平台,快速、高效地读取高质量的测序数据。
测序深度:PE 150
服务周期:90天
]RIP-seq技术参数
检测平台:通过Illumina测序,在全转录组范围内研究被特定蛋白特异结合的RNA区域或种类,且可比较多个样品间差异。
测序深度:PE 150 / SE 50
服务周期:60天
]服务内容
1.甲基化测序服务内容:
测序数据质量评估、与参考基因组比对、甲基化位点calling、甲基化分布、差异甲基化分析、相关基因分析、多样本分析。
2.RIP-seq服务内容:
测序数据质量评估、与参考基因组比对、peak峰calling、样品间相关性分析、motif分析、peak峰相关基因注释、差异peak分析、相关基因功能分析。
MAPQ (Mapping Quality),即比对质量值,表示reads比对到参考序列上某个位置的可靠程度,用错误比对到该位置的概率值来表示: MAPQ = -10log10(P),P是错误比对的概率。这里的错误比对,我们粗略的认为是非唯一比对。MAPQ越小,说明reads比对到该位置上的错误率越高。
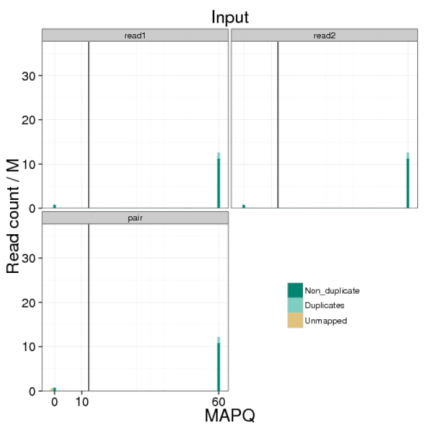
图1 比对质量分布图
样品间相关性分析是检验实验可靠性和样本选择是否合理的重要指标。相关系数越接近1,表明样品之间RNA甲基化模式的相似度越高。

图2 样品间相关性检查
对于单端测序,我们采用MACS2软件预测IP实验的片段大小(插入片段)。MACS以某个window size扫描基因组,统计每个window中read的富集程度,然后抽取1000个合适的window作样本构建富集模型,预测插入片段的长度。 对于双端测序,我们采用RSeQC软件对比对结果进行插入片段预测。利用预测得到的插入片段进行后续的peak分析。
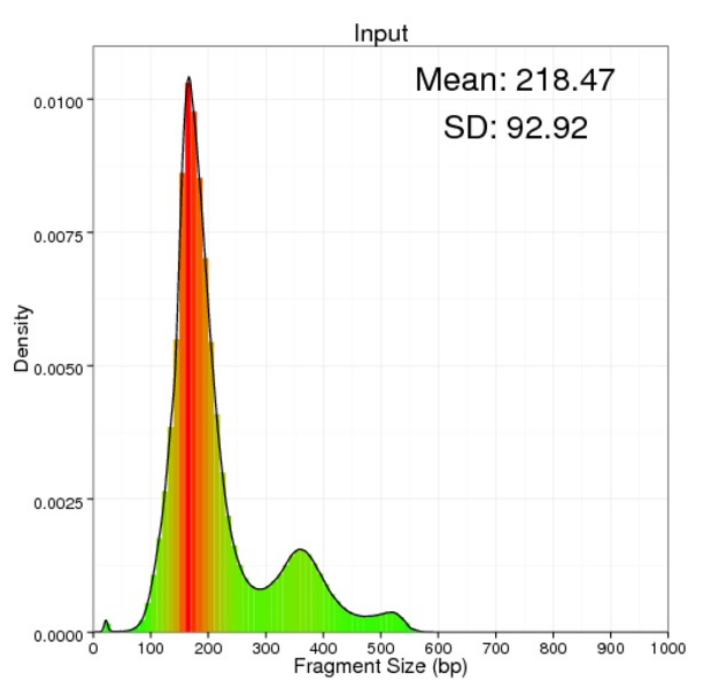
图3 插入片段长度分布图
随着新一代测序技术的发展,将m6A抗体富集后的RNA序列直接进行高通量测序,利用MACS2软件((阈值为qvalue=0.05)完成峰检分析(peak calling)并进行峰的个数、 宽度、分布等进行统计,以及筛选出峰的相关基因。
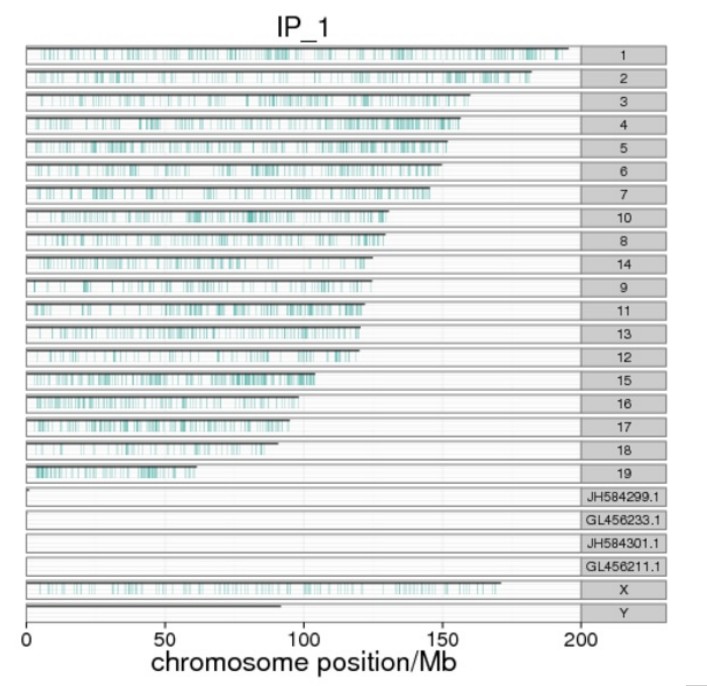

图4 峰在染色体上的分布 图5 峰的宽度分布
作为mRNA中最常见的甲基化修饰,m6A主要富集在mRNA的启动⼦子区、终⽌止密码子区,并且有特定的结合序列,通过结合到特定的位置上,从而在基因表达调控中发挥作用。我们利HOMER软件对MeRIP peak结合的mRNA区域上进行了motif (motif表示m6A位点的序列保守性)的识别。
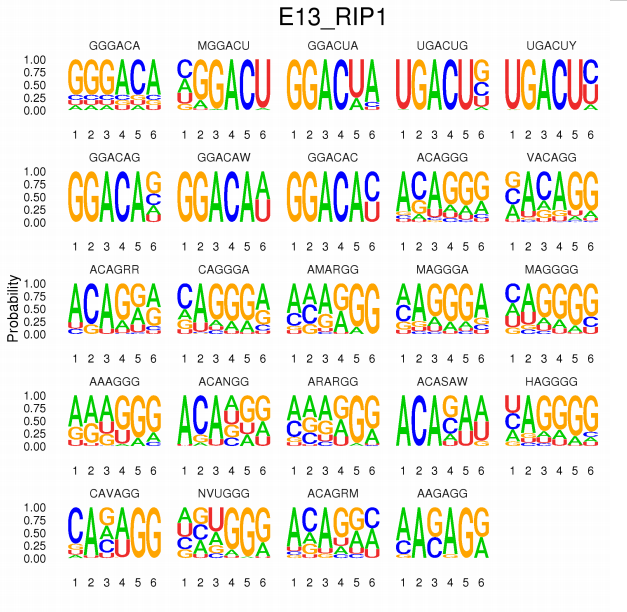
图6 motif序列
Peak 重叠基因GO富集柱状图,直观的反映出在生物过程(biological process)、 细胞组分(cellular component)和分子功能(molecular function)富集的GO项目上Peak 重叠基因的个数分布情况。
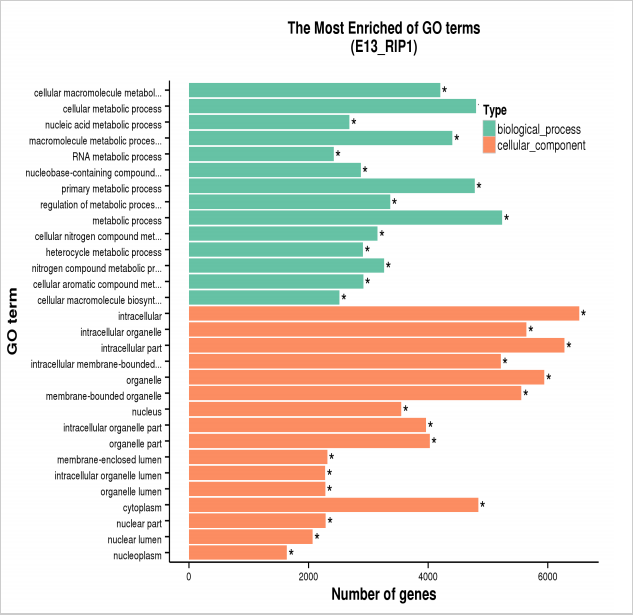
图7 GO富集分析
Peak 重叠基因KEGG富集散点图是KEGG富集分析结果的图形化展示方式。KEGG富集程度通过Rich factor、qvalue和富集到此通路上的基因个数来衡量。

图8 KEGG富集散点图
相关文献
Xu J, Zhou S, Gong X, et al. Single-base methylome analysis reveals dynamic epigenomic differences associated with water deficit in apple. Plant Biotechnol J. 2018;16(2):672-687.





